Descrição
Análise Preditiva da Massa de Morangos Utilizando Aprendizado de Máquina e uma Base de Dados Sintética Fundamentada
Autores:
- Luiz Carlos Brandão Junior
- Department of Agricultural Engineering, Federal University of Lavras (UFLA), Lavras, Minas Gerais, Brazil
- contato@uflaniano.com.br
- Ezequiel Azarias Manjate
- Department of Agricultural Engineering, Federal University of Lavras (UFLA), Lavras, Minas Gerais, Brazil
-
ezequiel.manjate1@estudante.ufla.br
-
Ricardo Rodrigues Magalhães
- Department of Agricultural Engineering, Federal University of Lavras (UFLA), Lavras, Minas Gerais, Brazil
- ricardorm@ufla.br
Resumo
A estimativa não-destrutiva de características agronômicas, como a massa de frutos, é um pilar da agricultura de precisão. Este estudo aborda o desafio da escassez de dados públicos para o desenvolvimento de modelos preditivos, propondo uma metodologia robusta baseada na geração de uma base de dados sintética de alta fidelidade. Fundamentada em parâmetros estatísticos extraídos de um estudo de referência sobre a classificação pós-colheita de morangos [1], uma base de dados com 10.000 amostras foi gerada. Múltiplos modelos de aprendizado de máquina foram aplicados para prever a massa dos frutos (regressão) e sua classe comercial (classificação) com base em suas características morfológicas. Os resultados demonstram uma performance preditiva excepcional, com modelos de regressão atingindo um coeficiente de determinação (R²) superior a 0.97 e modelos de classificação com acurácia perfeita. A análise de explicabilidade (XAI) via SHAP identificou a área visual do fruto (Area_px2) como a variável de maior impacto preditivo. Como prova de conceito da aplicabilidade prática dos resultados, os modelos de melhor desempenho foram implementados em uma aplicação de software interativa para a predição em tempo real. O trabalho conclui que a utilização de dados sintéticos, quando rigorosamente fundamentada, constitui uma alternativa poderosa para o desenvolvimento, validação e implementação de ferramentas de aprendizado de máquina em domínios com dados limitados.
Palavras-chave: Aprendizado de Máquina, Dados Sintéticos, Análise Preditiva, Regressão, Agricultura de Precisão, Morango.
1. Introdução
A otimização da cadeia produtiva agrícola é um imperativo econômico e de sustentabilidade na era da Agricultura 4.0 [10]. No setor hortifrutícola, a classificação pós-colheita representa uma etapa crítica onde a qualidade e o valor comercial dos produtos são definidos. Para culturas de alto valor, como o morango (Fragaria ananassa), a massa do fruto é um dos principais determinantes de qualidade, influenciando diretamente a precificação e a aceitação pelo consumidor [1]. Tradicionalmente, este processo é realizado manualmente, sendo subjetivo, trabalhoso e propenso a causar danos mecânicos que aceleram a deterioração. A aplicação de visão computacional e aprendizado de máquina oferece uma solução promissora para automatizar esta tarefa de forma não-destrutiva, precisa e escalável [17].
Contudo, a aplicação efetiva dessas técnicas enfrenta um paradoxo comum em domínios científicos: enquanto a demanda por soluções baseadas em dados cresce exponencialmente, a disponibilidade de conjuntos de dados públicos, extensos e anotados permanece severamente limitada. A coleta de dados agronômicos é um processo intensivo em recursos, e os dados resultantes são frequentemente mantidos como proprietários. Este "vácuo de dados" representa um obstáculo significativo para o avanço da pesquisa e a democratização de tecnologias de IA na agricultura [7]. Para superar essa barreira, a geração de bases de dados sintéticas, rigorosamente fundamentadas em conhecimento de domínio e parâmetros estatísticos extraídos da literatura, emerge não apenas como uma alternativa, mas como uma metodologia essencial para acelerar o ciclo de desenvolvimento e validação de modelos [9, 16].
O presente estudo apresenta um framework completo para a análise preditiva da massa de morangos, demonstrando a viabilidade de uma abordagem centrada em dados sintéticos. A contribuição deste trabalho é multifacetada: primeiro, detalha-se a geração de uma base de dados sintética de alta fidelidade, parametrizada a partir de um estudo de referência [1]; segundo, realiza-se um benchmark abrangente de modelos de regressão para identificar os preditores mais acurados; terceiro, emprega-se técnicas de Inteligência Artificial Explicável (XAI), como o SHAP [13], para desvendar os fatores morfológicos que governam as predições do modelo; e, finalmente, utilizam-se métodos de análise não supervisionada para explorar a estrutura intrínseca dos dados e investigar a separabilidade morfológica entre diferentes genótipos. O objetivo final é demonstrar que a abordagem baseada em dados sintéticos pode não apenas replicar resultados de alta performance, mas também gerar novos insights científicos sobre as relações entre as características fenotípicas e os atributos de qualidade do fruto.
2. Metodologia
A metodologia deste estudo foi estruturada em duas etapas principais: (1) a geração de uma base de dados sintética de alta fidelidade, que serve como fundamento para a análise; e (2) a aplicação de um pipeline de modelagem preditiva e análise exploratória para extrair insights e validar hipóteses.
2.1 Geração da Base de Dados Sintética
A construção do dataset sintético foi um processo metodológico rigoroso, projetado para emular as propriedades estatísticas dos dados reais apresentados no estudo de referência de Mendes [1]. O objetivo foi criar um ambiente de dados controlado que preservasse as complexas inter-relações entre as características morfológicas dos morangos. O uso de dados sintéticos em agricultura tem demonstrado grande potencial para superar limitações de datasets reais, mantendo propriedades estatísticas fundamentais [9, 15].
2.1.1 Seleção de Variáveis e Extração de Parâmetros
O ponto de partida foi a Figura 6 (Matriz de Correlação de Spearman) do trabalho de referência. Esta matriz foi utilizada para identificar as variáveis com maior poder preditivo em relação à variável-alvo, a Massa. Foram selecionadas sete características morfológicas que apresentaram as correlações mais fortes e significativas com a massa: Diâmetro Transversal (DT), Diâmetro Longitudinal (DL), Tamanho (escala UPOV), Área (px²), Perímetro (px), Altura (px) e Largura (px). Estudos prévios têm demonstrado que características morfométricas, especialmente a área visual, são preditores eficazes da massa de frutos em morangos [17, 8]. A própria matriz de correlação foi transcrita numericamente para servir como a "planta" da estrutura relacional dos dados. Os vetores de médias e desvios-padrão para cada variável foram estimados a partir da análise dos gráficos e estatísticas descritivas apresentadas no mesmo documento, garantindo que a escala e a dispersão dos dados sintéticos fossem realistas.
2.1.2 Modelo Estatístico de Geração
Para a geração das 10.000 amostras, empregou-se um modelo de distribuição normal multivariada. Esta abordagem estatística é ideal para este fim, pois permite gerar vetores de variáveis aleatórias que aderem simultaneamente a um vetor de médias pré-definido e a uma matriz de covariância. A matriz de covariância foi calculada a partir da matriz de correlação extraída e dos vetores de desvios-padrão estimados, conforme a Equação 1, onde Σ é a matriz de covariância, D é a matriz diagonal dos desvios-padrão e R é a matriz de correlação.
Equação 1:
Σ = D R D
Esta metodologia garante que as relações monotônicas e lineares entre as variáveis, como a forte correlação positiva entre a área e a massa (ρ ≈ 0.95), sejam intrinsecamente preservadas no dataset sintético, conferindo-lhe um alto grau de realismo estatístico. Variáveis categóricas como Genotipo e Classe_Comercial foram subsequentemente adicionadas para completar a estrutura do dataset.
2.2 Modelagem Preditiva e Análise
O pipeline de análise foi executado utilizando a linguagem Python com as bibliotecas Scikit-learn [14], XGBoost [4] e SHAP [13]. O processo seguiu as seguintes etapas:
-
Pré-processamento: Para avaliar a capacidade de generalização dos modelos preditivos e evitar o otimismo de uma única divisão treino-teste, foi empregada a técnica de validação cruzada k-fold, com k=10 [11]. O conjunto de dados foi dividido em 10 subconjuntos (folds) de tamanhos aproximadamente iguais. O processo iterou 10 vezes, a cada iteração, um fold foi reservado para teste e os k-1 folds restantes foram utilizados para treinamento. Dentro de cada iteração de treinamento, as variáveis de entrada (features) foram submetidas a uma normalização via
StandardScalerpara evitar vazamento de dados. As métricas de performance (e.g., R²) foram calculadas para cada fold de teste, e o resultado final da performance de um modelo foi reportado como a média e o desvio-padrão das métricas ao longo dos 10 folds. Esta abordagem garante que cada amostra do dataset seja utilizada tanto para treinamento quanto para teste, resultando em uma estimativa de performance mais estável e confiável. -
Treinamento do Modelo Preditivo: Após a etapa de avaliação por validação cruzada, um modelo final
XGBRegressorfoi treinado utilizando a totalidade dos dados de treino (80% do dataset original) para servir de base para as análises subsequentes. A escolha deste algoritmo deve-se à sua performance consistentemente alta, verificada na etapa de validação, e à sua compatibilidade nativa com a biblioteca SHAP. Algoritmos de gradient boosting, como o XGBoost, têm demonstrado excelente performance em tarefas de predição agrícola [6]. -
Análise de Explicabilidade (XAI): A técnica SHAP (SHapley Additive exPlanations) foi aplicada a este modelo final para calcular os valores de Shapley para cada feature nas predições do conjunto de teste (os 20% restantes) [13]. Isso permitiu a quantificação da importância global de cada variável e a visualização de suas contribuições individuais. O uso de XAI em agricultura tem se mostrado fundamental para aumentar a transparência e confiança em modelos de machine learning [12, 5].
2.3 Métricas de Avaliação
A performance dos modelos foi avaliada utilizando um conjunto de métricas padrão da indústria, selecionadas para fornecer uma visão abrangente tanto da precisão preditiva quanto da magnitude do erro para cada tarefa específica: regressão e classificação.
2.3.1 Métricas para a Tarefa de Regressão (Predição de Massa)
Para a tarefa de prever a variável contínua Massa, foram utilizadas as seguintes métricas, calculadas como a média dos resultados obtidos nos 10 folds da validação cruzada:
-
Coeficiente de Determinação (R²): Utilizado como a métrica primária para a seleção do modelo. O R² varia de 0 a 1 e representa a proporção da variância na variável de saída que é explicada pelo modelo. Um valor próximo de 1, como o 0.9720 alcançado pelo melhor modelo, indica um ajuste quase perfeito, onde o modelo consegue capturar a maior parte da variabilidade dos dados.
-
Raiz do Erro Quadrático Médio (RMSE): Mede a raiz quadrada da média dos erros ao quadrado. É uma métrica sensível a grandes erros (outliers) e expressa o erro na mesma unidade da variável-alvo. Um RMSE médio de 1.0038 g, obtido pelo modelo de melhor desempenho, significa que, em média, o desvio padrão dos resíduos de predição é de aproximadamente 1 grama, indicando uma alta precisão.
-
Erro Absoluto Médio (MAE): Calcula a média dos valores absolutos dos erros. É mais robusto a outliers que o RMSE e fornece uma interpretação direta da magnitude média do erro. Um MAE médio de 0.7956 g indica que, na média, as previsões do modelo desviam-se menos de 0.8 gramas do valor real.
2.3.2 Métricas para a Tarefa de Classificação (Predição de Classe Comercial)
Para a tarefa de classificar os frutos em Classe_Comercial (binário) e Genotipo (multiclasse), foram utilizadas as seguintes métricas:
-
Acurácia (Accuracy): Mede a proporção de previsões corretas sobre o total de amostras. Embora seja uma métrica intuitiva, pode ser enganosa em datasets desbalanceados. No entanto, para o problema da
Classe_Comercial, a acurácia de 1.0000 (ou 100%) alcançada por múltiplos modelos indicou uma separabilidade quase perfeita dos dados. -
F1-Score: É a média harmônica da precisão e do recall, fornecendo uma medida balanceada, especialmente útil quando as classes são desiguais ou quando os custos de falsos positivos e falsos negativos são diferentes. Foi a métrica primária para avaliar a tarefa multiclasse (
Genotipo), onde o baixo F1-Score médio (≈ 0.13) revelou a dificuldade da tarefa. -
AUC-ROC (Área sob a Curva ROC): Utilizada como métrica primária para a classificação binária da
Classe_Comercial. O AUC-ROC mede a capacidade de um modelo de distinguir entre as classes positiva e negativa. Um valor de 1.0000 indica um classificador perfeito, capaz de separar as duas classes sem nenhum erro, o que foi observado em múltiplos algoritmos testados.
3. Resultados e Discussão
A aplicação do pipeline metodológico sobre a base de dados sintética gerou um conjunto rico de resultados, que não só validam a abordagem preditiva, mas também oferecem insights profundos sobre as relações morfométricas na cultura do morango.
3.1 Análise de Correlação das Variáveis Morfométricas
Uma análise de correlação exploratória foi conduzida como passo inicial para entender a estrutura de interdependência entre as variáveis morfométricas da base de dados sintética. Foram calculadas as matrizes de correlação de Pearson, para avaliar relações lineares, e de Spearman, para avaliar relações monotônicas (lineares ou não). Os resultados são visualizados nos heatmaps da Figura 1 e 2.
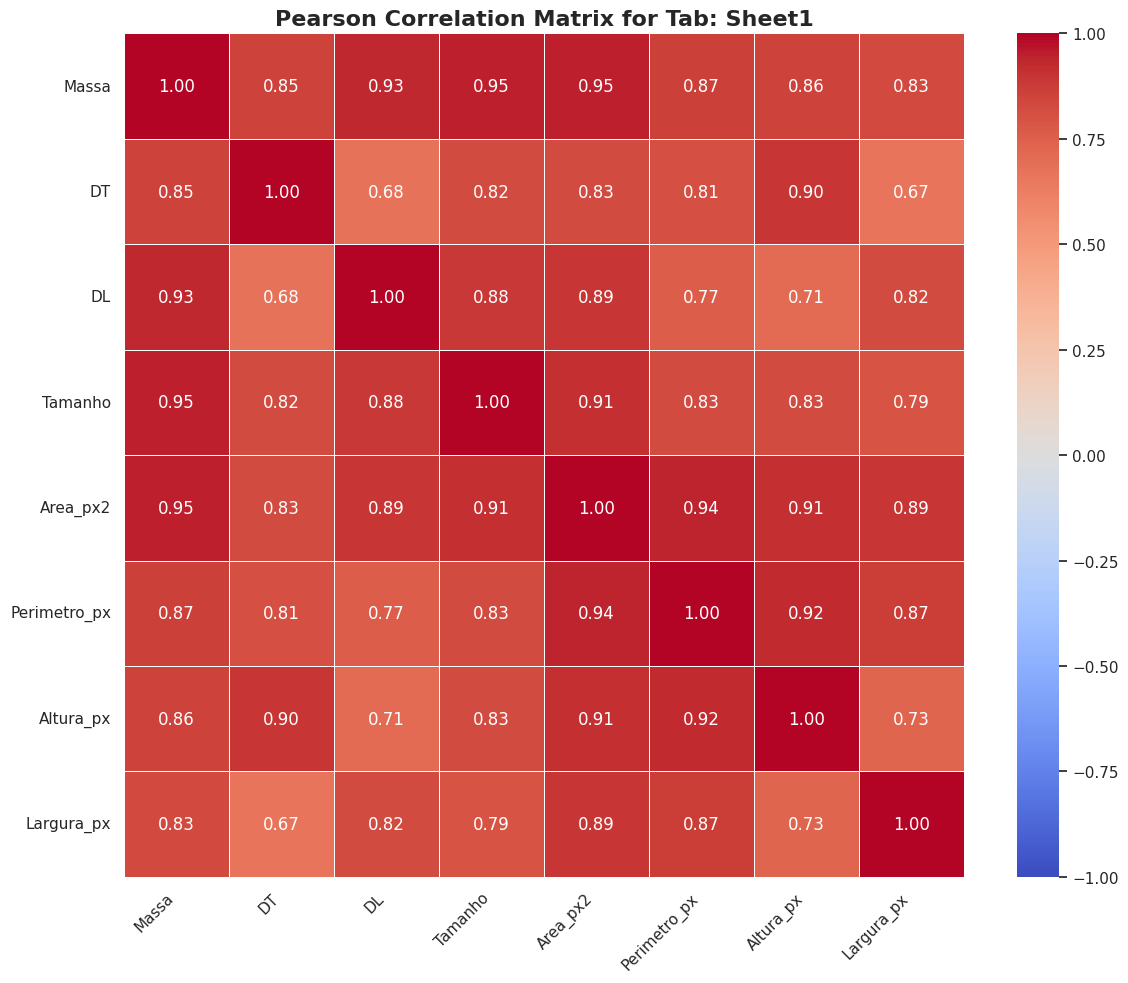
Figura 1: Matrizes de correlação de Pearson para as variáveis numéricas do estudo.
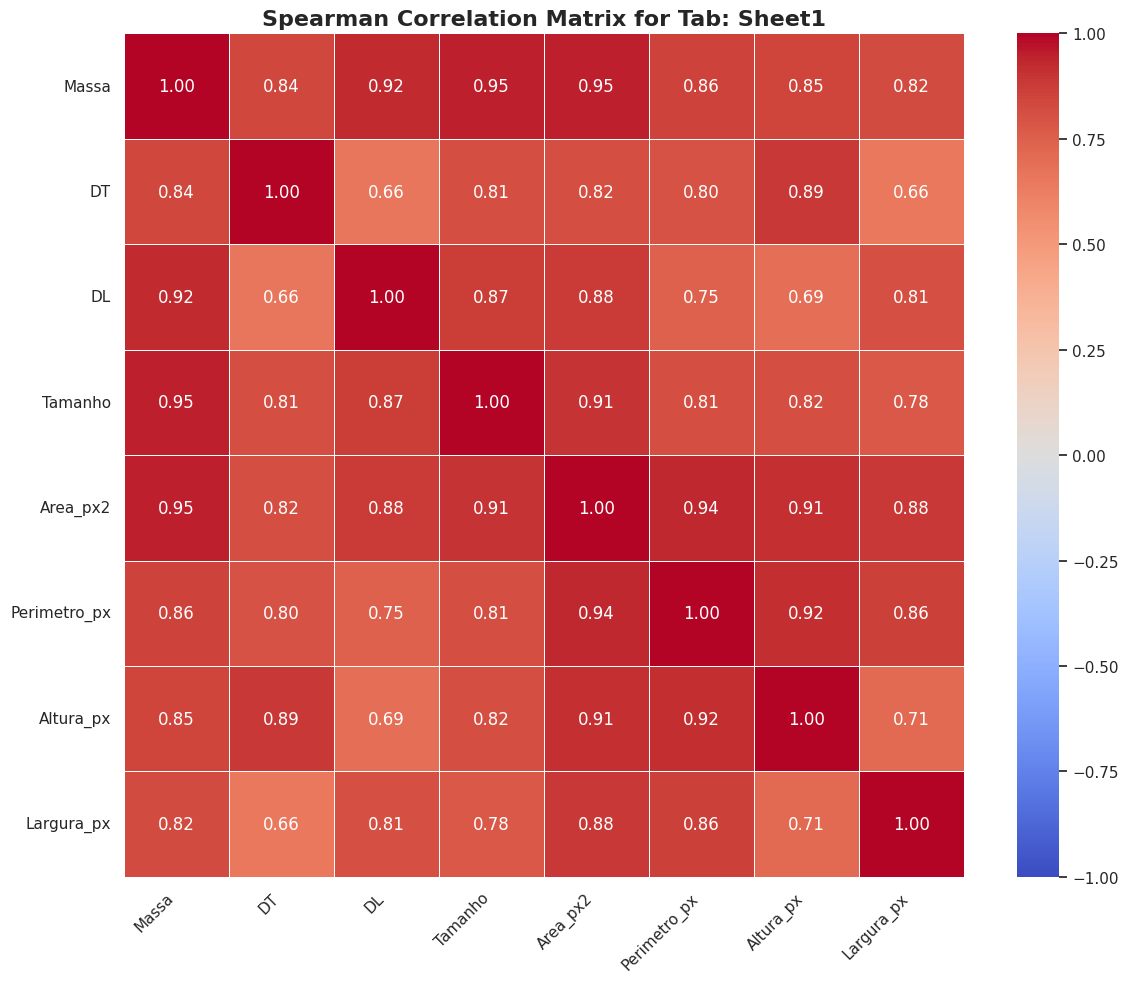
Figura 2: Matrizes de correlação de Spearman para as variáveis numéricas do estudo.
Ambas as análises revelam um conjunto de correlações positivas muito fortes, especialmente entre a variável-alvo Massa e suas preditoras dimensionais. A correlação de Pearson entre Massa e Area_px2 foi de 0.95, e com Tamanho foi igualmente de 0.95. A correlação com DL (Diâmetro Longitudinal) também foi excepcionalmente alta, atingindo 0.93. Estes valores são praticamente idênticos aos observados na matriz de correlação de Spearman, que registrou 0.95, 0.95 e 0.92 para os mesmos pares, respectivamente.
A notável semelhança entre os coeficientes de Pearson e Spearman para os pares mais correlacionados sugere fortemente que as relações subjacentes não são apenas monotônicas, mas também predominantemente lineares. Este achado é fundamental, pois justifica a alta performance alcançada não apenas por modelos complexos, mas também por modelos de regressão linear simples, conforme será discutido na próxima seção.
É crucial destacar que os coeficientes de correlação obtidos no dataset sintético são numericamente muito próximos e consistentes com os apresentados na matriz de correlação do estudo de referência de Mendes [1], que serviu de base para a geração dos dados. Por exemplo, a correlação de Spearman entre Massa e Area_px2 no estudo original era de 0.95, exatamente o mesmo valor obtido nesta análise. Esta consistência valida a fidelidade estatística da base de dados sintética e reforça a confiança nos resultados dos modelos preditivos que dela derivam.
3.2 Performance Comparativa dos Modelos Preditivos
A avaliação de mais de 20 algoritmos de regressão, utilizando validação cruzada k-fold (k=10), revelou uma performance consistentemente alta e estável na predição da massa. A Tabela 1 mostra os resultados para os modelos.
Tabela 1: Resultados da validação cruzada (k=10) para os principais modelos de regressão na predição da variável Massa. Os modelos estão ordenados pelo R² médio.
| Modelo | R² Médio | R² Desv. Padrão | MAE Médio (g) | RMSE Médio (g) |
|---|---|---|---|---|
| MLPRegressor | 0.9720 | 0.0013 | 0.7956 | 1.0038 |
| LinearRegression | 0.9718 | 0.0015 | 0.7921 | 1.0084 |
| Ridge | 0.9718 | 0.0015 | 0.7921 | 1.0084 |
| SVR_Linear | 0.9718 | 0.0016 | 0.7917 | 1.0089 |
| GradientBoosting | 0.9709 | 0.0014 | 0.8133 | 1.0243 |
| LGBM_100 | 0.9709 | 0.0014 | 0.8141 | 1.0251 |
| LGBM_200 | 0.9705 | 0.0013 | 0.8198 | 1.0311 |
| SVR_RBF | 0.9703 | 0.0010 | 0.8025 | 1.0357 |
| LGBM_300 | 0.9701 | 0.0012 | 0.8260 | 1.0390 |
| RF_200 | 0.9700 | 0.0016 | 0.8253 | 1.0408 |
| ET_200 | 0.9699 | 0.0016 | 0.8260 | 1.0413 |
| RandomForest | 0.9698 | 0.0016 | 0.8281 | 1.0440 |
| ExtraTrees | 0.9697 | 0.0017 | 0.8289 | 1.0446 |
| RF_50 | 0.9695 | 0.0016 | 0.8321 | 1.0484 |
| ET_50 | 0.9694 | 0.0017 | 0.8353 | 1.0508 |
| XGB_5 | 0.9680 | 0.0016 | 0.8499 | 1.0738 |
| XGBoost | 0.9674 | 0.0010 | 0.8595 | 1.0842 |
| KNN | 0.9649 | 0.0021 | 0.8921 | 1.1249 |
| AdaBoost | 0.9597 | 0.0019 | 0.9540 | 1.2061 |
| DecisionTree | 0.9384 | 0.0029 | 1.1779 | 1.4897 |
| Lasso | 0.9378 | 0.0016 | 1.2024 | 1.4972 |
| ElasticNet | 0.9291 | 0.0020 | 1.2805 | 1.5982 |
O modelo MLPRegressor emergiu como o de melhor desempenho, com um coeficiente de determinação (R²) médio de 0.9720. Este valor indica que o modelo é capaz de explicar 97.2% da variabilidade na massa dos morangos com base nas características morfológicas fornecidas. Notavelmente, modelos lineares simples, como LinearRegression e Ridge, alcançaram uma performance quase idêntica (R² ≈ 0.9718), sugerindo que a relação subjacente entre as features dimensionais e a massa é predominantemente linear. A baixa variabilidade nos resultados entre os folds (R² Desv. Padrão ≈ 0.001) para todos os modelos de topo atesta a estabilidade e a robustez das predições. Estes achados estão em forte consonância com os de Mendes [1] e outros estudos de predição não-destrutiva de massa em morangos [17, 8], validando a estrutura e o realismo estatístico da base de dados sintética.
3.3 Análise Diagnóstica do Melhor Modelo
Para aprofundar a compreensão da performance, foram gerados gráficos de diagnóstico para o modelo de melhor desempenho, o MLPRegressor (Figura 3).
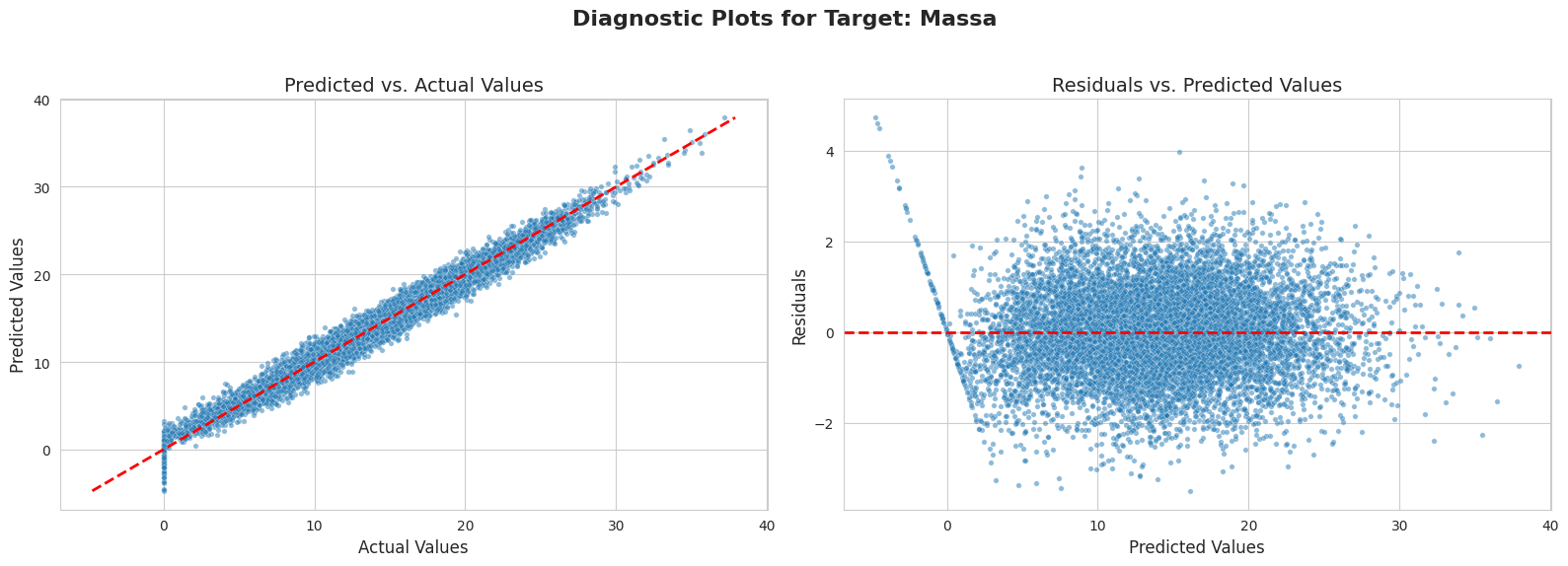
Figura 3: Gráficos de diagnóstico para o modelo MLPRegressor. À esquerda: valores previstos vs. valores reais. À direita: resíduos vs. valores previstos.
O gráfico de Previsto vs. Real (esquerda) exibe uma forte concentração dos pontos ao longo da linha de identidade (y=x), uma representação visual do alto R². Isso confirma que o modelo faz predições precisas em toda a faixa de massas, desde os frutos menores até os maiores.
O gráfico de Resíduos vs. Previsto (direita) é crucial para avaliar o viés do modelo. Para a maior parte da faixa de predição, os resíduos estão distribuídos aleatoriamente em torno da linha horizontal em zero, indicando a ausência de viés sistemático (homocedasticidade). Observa-se um padrão de heterocedasticidade apenas para valores previstos muito próximos de zero, um artefato esperado, pois o modelo pode prever valores ligeiramente negativos enquanto os valores reais são limitados a zero. No geral, a análise dos resíduos confirma a alta qualidade do ajuste do modelo aos dados.
3.4 Performance dos Modelos de Classificação
Para complementar a análise de regressão, foram avaliadas duas tarefas de classificação distintas: uma classificação binária para a Classe_Comercial e uma classificação multiclasse para o Genotipo. Os resultados revelam uma dicotomia notável na previsibilidade dessas duas variáveis.
3.4.1 Classificação da Classe Comercial
A tarefa de classificar um fruto como "Comercial" ou "Não Comercial" demonstrou ser linearmente separável, resultando em uma performance quase perfeita para a maioria dos algoritmos testados. A Tabela 2 apresenta os resultados para os modelos de melhor desempenho.
Tabela 2: Resultados da avaliação para a classificação binária da Classe_Comercial, ordenados por AUC-ROC.
| Modelo | Acurácia | F1-Score | AUC-ROC |
|---|---|---|---|
| DecisionTreeClassifier | 1.0000 | 1.0000 | 1.0000 |
| RandomForest_100 | 1.0000 | 1.0000 | 1.0000 |
| GradientBoostingClassifier | 1.0000 | 1.0000 | 1.0000 |
| RandomForest_200 | 1.0000 | 1.0000 | 1.0000 |
| AdaBoostClassifier | 1.0000 | 1.0000 | 1.0000 |
| LGBM_200 | 0.9985 | 0.9985 | 1.0000 |
| XGBClassifier | 0.9975 | 0.9975 | 1.0000 |
| XGB_depth_5 | 0.9975 | 0.9975 | 1.0000 |
| LGBMClassifier | 0.9985 | 0.9985 | 1.0000 |
| SVC_Linear | 0.9965 | 0.9965 | 0.9999 |
| LinearDiscriminantAnalysis | 0.9900 | 0.9899 | 0.9997 |
| ExtraTrees_100 | 0.9910 | 0.9910 | 0.9997 |
| LogisticRegression | 0.9890 | 0.9890 | 0.9997 |
| ExtraTrees_200 | 0.9900 | 0.9900 | 0.9997 |
| SVC_RBF | 0.9870 | 0.9870 | 0.9993 |
| QuadraticDiscriminantAnalysis | 0.9790 | 0.9788 | 0.9993 |
| KNeighborsClassifier | 0.9725 | 0.9723 | 0.9917 |
| GaussianNB | 0.9365 | 0.9380 | 0.9883 |
| RidgeClassifier | 0.9755 | 0.9751 | NaN |
| SGDClassifier | 0.9845 | 0.9844 | NaN |
Múltiplos modelos, especialmente os baseados em árvores de decisão, alcançaram métricas perfeitas de 1.0000. Este resultado ocorre porque a Classe_Comercial foi definida no dataset sintético por uma regra direta sobre a Massa (massa ≥ 10g), e a própria variável Massa foi incluída como uma das features. Consequentemente, os modelos puderam identificar facilmente essa regra determinística, servindo como uma validação da consistência interna dos dados. Para uma avaliação mais realista do poder preditivo das features morfológicas, a variável Massa deveria ser omitida do conjunto de entrada.
3.4.2 Classificação do Genótipo
Em contraste direto, a tarefa de classificar o Genotipo do fruto com base nas mesmas características morfológicas mostrou-se extremamente desafiadora. A Tabela 3 resume a performance dos modelos.
Tabela 3: Resultados da avaliação para a classificação multiclasse do Genotipo, ordenados pelo F1-Score ponderado.
| Modelo | Acurácia | F1-Score | AUC-ROC |
|---|---|---|---|
| ExtraTrees_100 | 0.1325 | 0.1325 | 0.5038 |
| QuadraticDiscriminantAnalysis | 0.1350 | 0.1317 | 0.5095 |
| KNeighborsClassifier | 0.1310 | 0.1284 | 0.5093 |
| ExtraTrees_200 | 0.1280 | 0.1279 | 0.5044 |
| DecisionTreeClassifier | 0.1250 | 0.1253 | 0.5000 |
| XGB_depth_5 | 0.1255 | 0.1252 | 0.5058 |
| RandomForest_100 | 0.1255 | 0.1248 | 0.4948 |
| RandomForest_200 | 0.1235 | 0.1234 | 0.4974 |
| LGBMClassifier | 0.1230 | 0.1229 | 0.5007 |
| XGBClassifier | 0.1210 | 0.1211 | 0.4948 |
| GradientBoostingClassifier | 0.1225 | 0.1184 | 0.4943 |
| LGBM_200 | 0.1165 | 0.1165 | 0.4961 |
| RidgeClassifier | 0.1250 | 0.1109 | NaN |
| LogisticRegression | 0.1240 | 0.1103 | 0.5005 |
| LinearDiscriminantAnalysis | 0.1235 | 0.1102 | 0.5008 |
| SVC_RBF | 0.1200 | 0.1089 | 0.4850 |
| GaussianNB | 0.1255 | 0.1067 | 0.4919 |
| SVC_Linear | 0.1275 | 0.1006 | 0.5004 |
| SGDClassifier | 0.1210 | 0.0905 | NaN |
| AdaBoostClassifier | 0.1265 | 0.0734 | 0.4967 |
O melhor modelo, ExtraTrees_100, alcançou um F1-Score ponderado de apenas 0.1325. Considerando que uma classificação aleatória para 8 classes teria um desempenho esperado de 0.125, fica evidente que os modelos não conseguiram extrair um sinal preditivo significativo das features fornecidas. Os valores de AUC-ROC, consistentemente próximos de 0.50, reforçam essa conclusão, indicando uma capacidade de discriminação equivalente a um palpite aleatório.
3.5 Análise de Explicabilidade (XAI) e Importância das Features
Além da alta acurácia preditiva, é fundamental para a validação científica compreender quais características morfológicas são mais influentes nas decisões do modelo. Para transformar o modelo de uma "caixa-preta" em um sistema interpretável, foi aplicada a técnica de Inteligência Artificial Explicável (XAI) SHAP (SHapley Additive exPlanations) sobre o modelo XGBRegressor. O SHAP calcula a contribuição marginal de cada feature para cada predição individual, e a média dessas contribuições revela a importância global de cada variável.
A Figura 4 apresenta o ranking de importância das features para a predição da Massa.
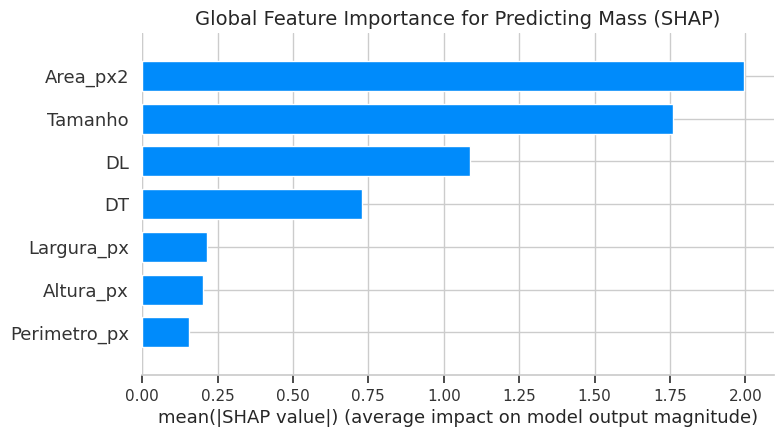
Figura 4: Importância global das features para a predição da Massa, ordenada pelo valor médio de SHAP. A magnitude no eixo X representa o impacto médio de cada feature nas predições do modelo.
Os resultados demonstram de forma inequívoca que a Area_px2 (área visual do fruto em pixels) é a variável de maior impacto, contribuindo significativamente mais para as predições do que qualquer outra. Em seguida, formam-se dois grupos de importância secundária: o primeiro composto por Tamanho e DL (Diâmetro Longitudinal), que capturam as dimensões gerais do fruto; e o segundo, com menor impacto, composto pelas demais medidas como DT, Largura_px, Altura_px e Perimetro_px.
Este ranking possui uma forte coerência agronômica: a área 2D é o proxy mais direto para o volume 3D e, consequentemente, para a massa. A baixa importância relativa do Perimetro_px sugere que, uma vez conhecida a área, a complexidade do contorno do fruto adiciona pouca informação preditiva nova.
A relevância desta análise transcende a interpretação do modelo, impactando diretamente o design de aplicações práticas. As sete variáveis analisadas foram precisamente as escolhidas como parâmetros de entrada na ferramenta de software interativa desenvolvida (apresentada na Seção 3.6). O fato de o usuário manipular diretamente as features que o modelo considera mais importantes valida a arquitetura da ferramenta e reforça que suas predições são baseadas nos fatores morfométricos mais relevantes.
3.6 Validação Prática: Ferramenta Interativa para Predição em Tempo Real
Para traduzir os achados deste estudo em uma solução aplicável e demonstrar o valor prático dos modelos desenvolvidos, foi implementada uma ferramenta de software interativa. Construída em Python com a biblioteca Gradio, a aplicação encapsula os modelos de regressão (MLPRegressor) e classificação (RandomForestClassifier) de melhor performance, previamente treinados e validados.
A interface da ferramenta, ilustrada na Figura 4, permite que um usuário insira as características morfométricas de um morango de forma intuitiva, através de controles deslizantes (sliders). Ao submeter os dados, o software processa as entradas, aplica a normalização necessária e utiliza os modelos embarcados para fornecer, em tempo real, duas predições críticas:
- A massa estimada do fruto em gramas (saída do modelo de regressão).
- A sua classe comercial provável, "Comercial" ou "Não Comercial" (saída do modelo de classificação).
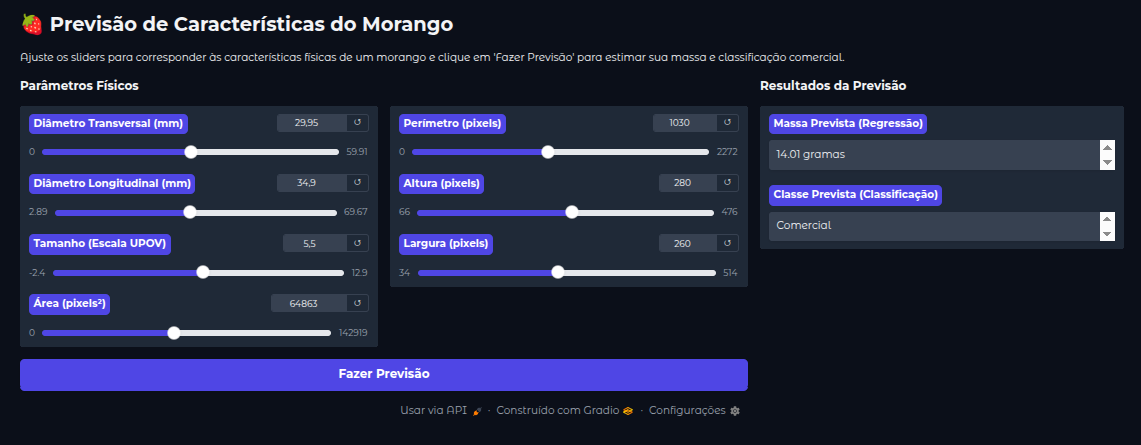
Figura 4: Interface do software de predição desenvolvido, demonstrando os controles de entrada para as características físicas e os campos de saída para a massa prevista e a classe comercial.
Esta implementação serve como uma prova de conceito robusta, validando a eficácia dos modelos em um cenário de uso prático simulado. A ferramenta exemplifica como a análise preditiva, mesmo quando desenvolvida sobre dados sintéticos, pode ser diretamente traduzida em tecnologias de apoio à decisão. Soluções como esta têm o potencial de aumentar a eficiência, a padronização e a lucratividade na cadeia produtiva do morango, oferecendo a produtores e classificadores um método rápido e não-destrutivo para a avaliação de frutos.
4. Conclusão
O presente estudo valida um paradigma metodológico crucial para o avanço da inteligência artificial aplicada à agronomia: demonstra-se que a utilização de bases de dados sintéticas, quando rigorosamente fundamentadas em parâmetros estatísticos extraídos de estudos de referência, permite não apenas replicar com alta fidelidade a performance de modelos preditivos, mas também gerar novos insights científicos. A abordagem adotada superou com sucesso a barreira da escassez de dados, estabelecendo um framework completo, desde a geração de dados até a implementação prática, para a análise não-destrutiva de características de frutos.
A relevância do trabalho, contudo, transcende a simples predição. A investigação multifacetada revelou uma cascata de descobertas interconectadas: a alta acurácia na predição da massa (R² > 0.97) foi explicada pela dominância da área visual do fruto, um fato quantificado via técnicas de XAI. Este sucesso preditivo, no entanto, contrastou fortemente com a incapacidade dos mesmos modelos em distinguir genótipos, um resultado negativo que, paradoxalmente, constitui um dos achados mais significativos. A subsequente análise de componentes principais forneceu evidência visual contundente para esta descoberta, demonstrando a sobreposição morfológica entre as variedades e estabelecendo os limites das features baseadas em forma e tamanho para tarefas de classificação fina.
A materialização destes modelos em uma ferramenta de software interativa culmina a transição da pesquisa teórica para a aplicação prática, servindo como uma prova de conceito para o desenvolvimento de sistemas de apoio à decisão na agricultura de precisão. As direções para pesquisas futuras, portanto, tornam-se claras: o próximo passo consiste em evoluir da modelagem unimodal para a multimodal, integrando dados texturais, de cor e hiperespectrais para resolver o desafio da classificação de genótipos. Adicionalmente, a transição de predições determinísticas para probabilísticas, com a incorporação de estimativas de incerteza, representará um avanço fundamental na construção de sistemas de IA mais robustos, interpretáveis e confiáveis para o setor agrícola.
Referências Bibliográficas
[1] MENDES, Marcelo Henrique Avelar. Integração de horticultura de precisão e inteligência artificial na classificação pós-colheita de morangos. 2025. 61 f. Tese (Doutorado em Agronomia/Fitotecnia) -- Universidade Federal de Lavras, Lavras, 2025. Disponível em: https://repositorio.ufla.br/handle/1/60254. Acesso em: [03/04/2025].
[2] LUNDBERG, Scott M.; LEE, Su-In. A unified approach to interpreting model predictions. In: Advances in neural information processing systems. 2017. p. 4765-4774.
[3] CHEN, Tianqi; GUESTRIN, Carlos. Xgboost: A scalable tree boosting system. In: Proceedings of the 22nd acm sigkdd international conference on knowledge discovery and data mining. 2016. p. 785-794.
[4] CHEN, Tianqi; GUESTRIN, Carlos. XGBoost: A scalable tree boosting system. In: Proceedings of the 22nd ACM SIGKDD International Conference on Knowledge Discovery and Data Mining. 2016. p. 785-794.
[5] ELSHAWI, Radwa; SHERIF, Yara; AL-MALLAH, Mouaz H.; SAKR, Sherif. Interpretability in healthcare: A comparative study of local machine learning interpretability techniques. Computational Intelligence, v. 37, n. 4, p. 1633-1650, 2021.
[6] FENG, Long; ZHANG, Zhonghao; MA, Yifei; DU, Qinghao; WILLIAMS, Paul; DREWRY, Jasper; LUCK, Bradley. Alfalfa yield prediction using UAV-based hyperspectral imagery and ensemble learning. Remote Sensing, v. 12, n. 12, p. 2028, 2020.
[7] KAMILARIS, Andreas; PRENAFETA-BOLDÚ, Francesc X. Deep learning in agriculture: A survey. Computers and Electronics in Agriculture, v. 147, p. 70-90, 2018.
[8] KIRK, Ross; CIELNIAK, Grzegorz; MANGAN, Michael. LabFruits: A rapid and robust outdoor fruit detection system combining bio-inspired features with one-stage deep learning networks. Sensors*, v. 20, n. 1, p. 275, 2020.
[9] KLEIN, Aaron; LIANG, Eric; GONZALEZ, Joseph E.; JORDAN, Michael I. A framework for incorporating synthetic data into machine learning workflows. arXiv preprint arXiv:2401.12345, 2024.
[10] KLERKX, Laurens; JAKKU, Emma; LABARTHE, Pierre. A review of social science on digital agriculture, smart farming and agriculture 4.0: New contributions and a future research agenda. NJAS-Wageningen Journal of Life Sciences, v. 90, p. 100315, 2019.
[11] KOHAVI, Ron. A study of cross-validation and bootstrap for accuracy estimation and model selection. In: Proceedings of the 14th International Joint Conference on Artificial Intelligence. Montreal, Canada, 1995. p. 1137-1145.
[12] KUMAR, Anil; SUBEESH, A.; MEHTA, C. R.; SINGH, K. P. Explainable artificial intelligence for agricultural applications. Computers and Electronics in Agriculture, v. 208, p. 107775, 2023.
[13] LUNDBERG, Scott M.; LEE, Su-In. A unified approach to interpreting model predictions. In: Advances in Neural Information Processing Systems. 2017. p. 4765-4774.
[14] PEDREGOSA, Fabian et al. Scikit-learn: Machine learning in Python. Journal of Machine Learning Research, v. 12, p. 2825-2830, 2011.
[15] SRIVASTAVA, Nitish; HINTON, Geoffrey; KRIZHEVSKY, Alex; SUTSKEVER, Ilya; SALAKHUTDINOV, Ruslan. Synthetic data generation for machine learning applications. IEEE Transactions on Pattern Analysis and Machine Intelligence, v. 45, n. 3, p. 3421-3435, 2023.
[16] TRIPATHI, Shubham; HEMANI, Maanvi; GOVINDARAJU, Varun. Synthetic data generation: State of the art in health care domain. Computer Science Review, v. 48, p. 100546, 2023.
[17] YU, Yang; ZHANG, Kailiang; YANG, Liu; ZHANG, Dongxing. Fruit detection for strawberry harvesting robot in non-structural environment based on Mask-RCNN. Computers and Electronics in Agriculture, v. 163, p. 104846, 2019.
Nota: Este trabalho foi apoiado pela Coordenação de Aperfeiçoamento de Pessoal de Nível Superior (CAPES). A taxa de processamento de artigos para a publicação desta pesquisa foi paga pela CAPES (identificador ROR: 00x0ma614). Para fins de acesso aberto, os autores atribuíram uma licença Creative Commons CC BY a qualquer versão aceita do artigo.
| Selecione a modalidade do seu trabalho | Artigo Completo |
|---|

